Índice:
- Etapa 1: Organização de dados de mamografia
- Etapa 2: processamento de imagem
- Etapa 3: Código de Limiar
- Etapa 4: Encontrando Anormalidades para Cada Imagem Binária
- Etapa 5: traçar a localização e o tamanho da massa diagnosticada para comparação visual
- Etapa 6: Implementando o Segundo Método de Comparação
- Etapa 7: Analisando os dados coletados
- Etapa 8: Fazendo seu próprio classificador
- Etapa 9: melhorias? Alguma ideia?
- Autor John Day day@howwhatproduce.com.
- Public 2024-01-30 11:37.
- Última modificação 2025-01-23 15:03.
O objetivo deste projeto foi identificar e usar um parâmetro para processar imagens de mamografia em tons de cinza de várias classificações de tecido de fundo: Tecido adiposo, glandular gorduroso e tecido denso. Essa classificação é usada quando os radiologistas analisam mamografias e precisam considerar se a densidade do tecido irá obscurecer quaisquer anormalidades, como lesões ou tumores. Isso ocorre porque ambas as estruturas fisiológicas normais, como tecido glandular e tecido conjuntivo fibroso. e morfologias anormais, como calcificações e tumores, aparecerão muito brilhantes na mamografia, enquanto o tecido adiposo menos denso aparecerá preto. Portanto, foi apropriado programar um classificador que possa manipular os níveis de intensidade de pixel para melhor visualizar e identificar as massas.
Etapa 1: Organização de dados de mamografia
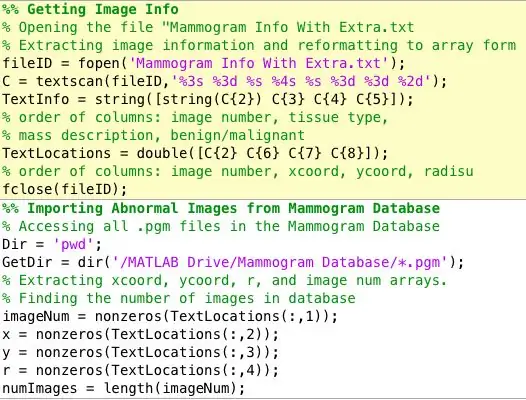
Uma das primeiras coisas que percebi que precisava lidar foi organizar os dados de uma forma muito clara, concisa e acessível. Estas são as variáveis que extraí da base de dados mini-MIAS de mamografias. Eu criei duas matrizes. Um contendo 4 colunas:
- Número da imagem:
- coordenada x de massa
- coordenada y de massa
- Raio de massa: (Isso definiu um tamanho aproximado para a massa
A segunda matriz continha informações de classificação:
- Tipo de tecido de fundo: gordo (F), glandular gorduroso (G), denso (D)
- Descrição da Massa: Bem definida (CIRC), espiculada (SPIC), outra mal definida (MISC) Distorção arquitetônica (ARCH), Assimetria (ASYM), Normal (NORM)
- Diagnósticos: Benigno (B), Maligno (M)
Como o objetivo deste projeto era determinar o melhor limite para cada tipo de tecido de fundo, nem todas as informações eram necessárias. No entanto, você pode expandir seu projeto para incluir análise de textura e testar seu classificador em relação às descrições de massa conhecidas.
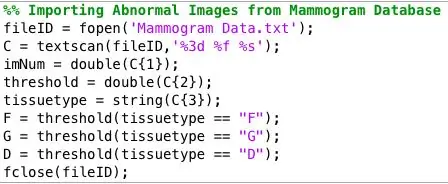
Nota lateral: O banco de dados de onde obtive as imagens de mamografia diagnosticadas organizou as informações sobre cada mamografia em um arquivo de texto separado das imagens. Foi um tanto difícil para mim extrair os dados de um arquivo de texto e organizá-los em formatos de matriz, mas o link a seguir foi muito útil para descobrir tudo isso. Como alternativa, apenas ajuste o código que colei acima para seus propósitos.
Formato de arquivo de mamografia: mdb001 G CIRC B 535 425 197
mdb002 G CIRC B 522 280 69
Ajuda do TextScan: https://www.mathworks.com/help/matlab/ref/textsca…Mammogram Database:
Etapa 2: processamento de imagem
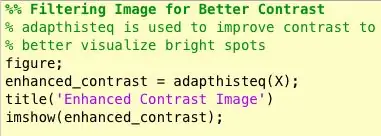
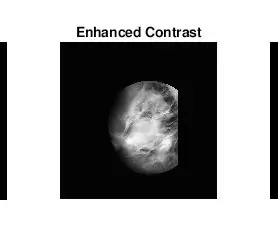
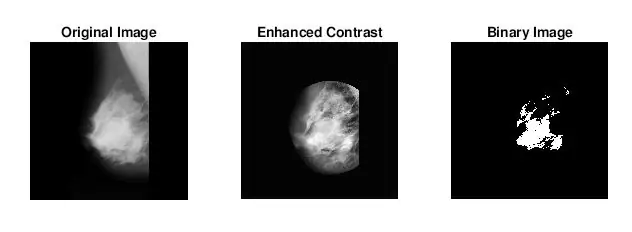
Bem, a segunda coisa que surgiu quando eu estava descobrindo como identificar massas foi que, para muitas mamografias anormais, eu não conseguia dizer visualmente onde a anormalidade estava ou quão grande ela era. Obviamente, como não sou um radiologista experiente, era de se esperar. No entanto, a maneira mais direta de encontrar anormalidades (de acordo com minhas longas pesquisas no Google) era observar as concentrações de áreas claras e escuras. Usei principalmente a função adapthisteq para aumentar o contraste da imagem e, em seguida, embinarizar para converter a imagem em uma imagem binária para experimentar diferentes níveis de limiar.
- adapthisteq: Esta função transforma os valores de intensidade de imagens em tons de cinza e rgb usando equalização de histograma adaptativa limitada por contraste. Em outras palavras, ele ajusta o histograma dos valores de intensidade para um tipo específico de distribuição. O link de matemática para esta função está anexado abaixo para leitura adicional.
- imbinarizar: cria uma imagem binária a partir de uma imagem em escala de cinza atribuindo todos os pixels acima de uma certa intensidade a 1s e os pixels abaixo desse valor a 0. Usei essa função para testar o limite ideal para reduzir o ruído do tecido de fundo.
Etapa 3: Código de Limiar
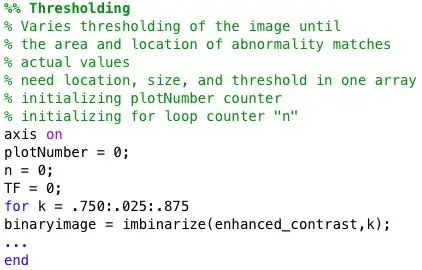
Um loop for é usado para binarizar a mamografia com limites variados. Para dar uma visão mais ampla, o loop for contém o código da Etapa 3 à Etapa 7. Assim, cada imagem binária será analisada quanto a anormalidades. Além disso, esse loop for está encerrado em outro loop for que importa uma nova imagem de mamografia do banco de dados a cada iteração.
Etapa 4: Encontrando Anormalidades para Cada Imagem Binária
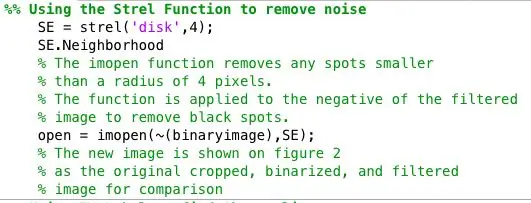
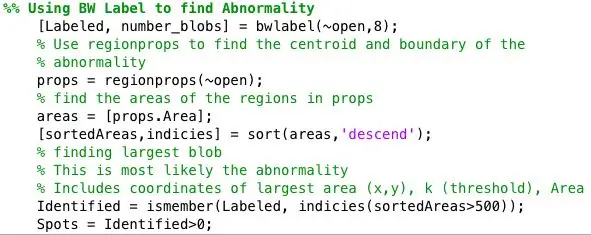
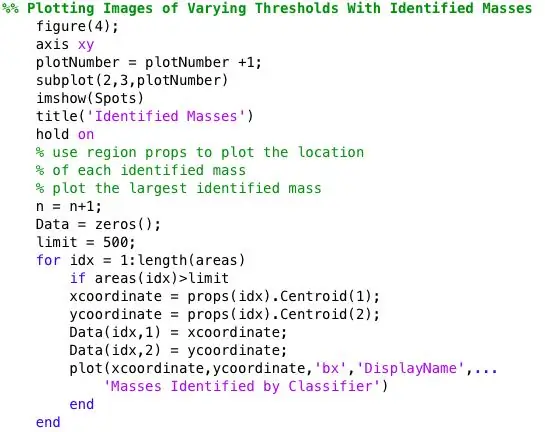
Processei posteriormente as imagens binárias usando a função strel em conjunto com imopen para remover o ruído de fundo. A imagem binária da etapa anterior é invertida e filtrada usando a vizinhança definida por SE. Então usei bwlabel para rotular todas as áreas que tinham pelo menos 8 pixels conectados.
A função region props foi usada para encontrar as propriedades do centroide e da área de cada ponto identificado por bwlabel.
Em seguida, todos os pontos maiores do que 500 pixels foram identificados usando ismember. Os centróides para os pontos identificados foram plotados em uma imagem que exibia apenas os pontos com área maior que 500. Área identificada = ismember (etiquetada, indicies (SortAreas> 500)); Pontos = Identificados> 0;
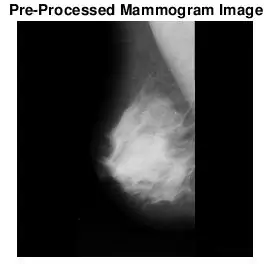
Etapa 5: traçar a localização e o tamanho da massa diagnosticada para comparação visual
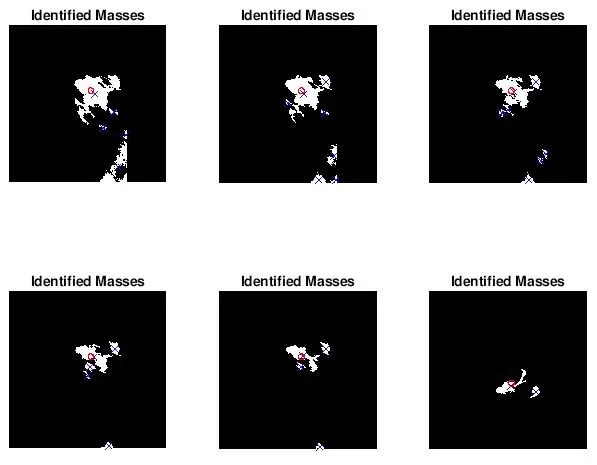

Queria ver se os pontos encontrados por bwlabel estavam corretos. Eu fiz isso de duas maneiras. Primeiro analisei a precisão do meu classificador fazendo uma comparação visual. Eu simplesmente plotei o tamanho real e a localização da anormalidade (círculo vermelho) e a localização determinada pelo código (x azul) na imagem da mamografia pré-processada. As seis imagens acima mostram os efeitos do aumento do valor do limite da escala de cinza.
Etapa 6: Implementando o Segundo Método de Comparação

A segunda maneira de testar o classificador e os valores de limite foi determinando se os locais encontrados pelo classificador estavam a uma certa distância das coordenadas de anomalia diagnosticadas. Salvei os limites para os quais pelo menos um dos pontos identificados estava dentro de 1,5 * r da anormalidade conhecida em um arquivo de texto separado chamado Dados de mamografia. O objetivo era encontrar o limite mínimo necessário para que meu classificador identificasse a anormalidade.
Etapa 7: Analisando os dados coletados
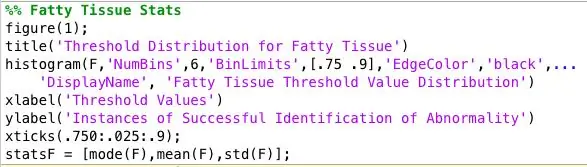
Eu executei o programa em todas as imagens anormais de mamografia e fiquei com um enorme arquivo de texto de dados. Para encontrar o melhor limite para cada tipo de tecido, organizei os dados por tipo de tecido e plotei um histograma dos valores de limite para cada tipo de tecido. O valor de limite adequado foi decidido com base em qual limite forneceu os resultados mais precisos para cada tipo de tecido. Salvei esses dados para fazer upload para meu classificador.
Etapa 8: Fazendo seu próprio classificador

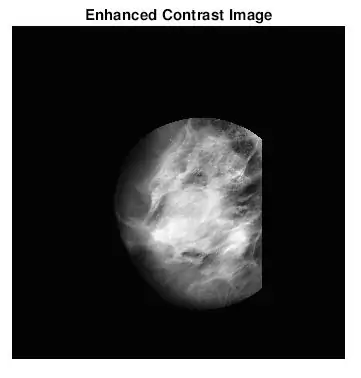
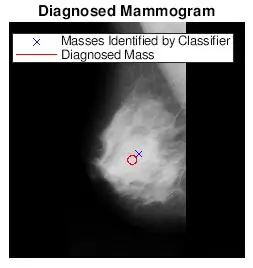
Depois de encontrar os valores de limite mais adequados para cada tipo de tecido, editei meu código original para que um usuário insira o número da imagem e o tipo de tecido para escolher o limite para a imagem da mamografia. Em seguida, tracei a localização da mamografia diagnosticada com as localizações encontradas nas imagens originais da mamografia. Eu queria tornar isso mais divertido, então programei uma função para cortar uma região circular em torno do ROI. O usuário seria instruído a escolher um ponto central e vários pontos que melhor englobassem o ROI. Anexei os dois arquivos matlab aqui.
Etapa 9: melhorias? Alguma ideia?
Enquanto escrevia este instrutível, comecei a ver muitas melhorias que poderia fazer no classificador, como encontrar maneiras de distinguir entre diferentes tipos de massas identificadas com base na análise de textura ou melhorar meus testes de seção de precisão do SandBoxProject. Arquivo. Como este era um projeto com prazo, tive que parar em algum lugar, mas espero poder usar as habilidades de processamento de imagens que aprendi em outros aplicativos. Além disso, anexei o arquivo que foi usado para processar em lote todas as imagens de mamografia anormais.
Recomendado:
Links de variáveis em Twine With Sugarcube: 10 etapas

Links de variáveis em Twine With Sugarcube: Estou tão feliz que você se juntou a mim novamente! Isso significa que o Wumpus ainda não o comeu. Bom! Para aqueles de vocês que não estão familiarizados, este é um conjunto de tutoriais que estou fazendo para ajudar minha adorável prima widdle oochy-skootchy com seu projeto de último ano. Este tutorial
Melhor maneira de visualizar / documentar arquivos em instructables: 4 etapas
Melhor maneira de visualizar / documentar arquivos em instructables: as pessoas costumam incluir seus arquivos para projeto por meio de uploads de instructables. Freqüentemente, são arquivos de origem de software que podem até mesmo ser o coração pulsante do instructable. Mas o Instructables não oferece ao leitor uma maneira fácil de ler e revisar o código. (
Medidor de intensidade de luz sem programação: 7 etapas (com imagens)

Medidor de intensidade de luz sem programação: Este instrutível é sobre como fazer um medidor de intensidade de luz básico sem usar Arduino ou qualquer outro microcontrolador ou programação. O medidor de intensidade de luz exibe diferentes níveis de intensidade de luz com diferentes cores de LEDs. O LED vermelho
Como voltar à interface original para visualizar os instrutíveis: 3 etapas

Como voltar para a interface original para visualizar os Instructables: Se você não percebeu, a interface do Instructables para visualizar as coisas foi alterada para destaque, popular, classificação | recente, visualizações e zeitgeist. Eu vi algumas reclamações de que eles não gostaram dessa mudança, então para todos: aqui é onde nós mudamos
N: Como fazer uma escultura em acrílico multicamadas e LED com níveis de iluminação variáveis: 11 etapas (com imagens)

N: Como fazer uma escultura em acrílico multicamadas e LED com níveis de iluminação variáveis: Aqui você pode descobrir como fazer você mesmo n feito para a exposição www.laplandscape.co.uk com curadoria do grupo de arte / design Lapland. Mais imagens podem ser vistas no flickrEsta exposição decorre de quarta-feira, 26 de novembro a sexta-feira, 12 de dezembro de 2008, inclusive
