Índice:
- Etapa 1: usando o Brain Box
- Etapa 2: Introdução: Analisando as seções transversais
- Etapa 3: Configurando uma função: LevelCurveTracings.m
- Etapa 4: desenvolvendo o algoritmo de limite: encontrando limites
- Etapa 5: Desenvolvendo o Algoritmo Bound: Gerando Matriz de Limite Externo
- Etapa 6: Desenvolvendo o Algoritmo Bound: Trabalhando com o Ponto Central
- Etapa 7: Desenvolvendo o algoritmo vinculado: quando um centróide não está centrado
- Etapa 8: Desenvolvendo o Algoritmo Bound: Interferência de Orifícios
- Etapa 9: desenvolvendo o algoritmo de limite: localizando buracos, finalizando limites do cérebro e limites de buracos
- Etapa 10: Registro de dados: Function PatientFiles.m
- Etapa 11: registro de dados em arquivos
- Etapa 12: Registro de dados: exibição de um gráfico do volume do cérebro ao longo do tempo
- Etapa 13: Fechando lacunas em subtramas: Subplotclose.m
- Etapa 14: O código principal: limpar tudo e solicitar entradas
- Etapa 15: O código principal: processamento em lote das imagens
- Etapa 16: O Código Principal: Preenchimento
- Etapa 17: O Código Principal: Determinando Limites
- Etapa 18: O Código Principal: Determinando a Dimensão Z Adequada
- Etapa 19: O Código Principal: Determinando as Coordenadas X e Y
- Etapa 20: O código principal: traçando uma estrutura tridimensional, encontrando o volume e registrando os dados
- Autor John Day day@howwhatproduce.com.
- Public 2024-01-31 10:23.
- Última modificação 2025-01-23 15:03.
O avanço na fronteira de uma vida humana mais longa trouxe o surgimento de doenças não vistas por civilizações antes da nossa. Entre estes, o Alzheimer afetou aproximadamente 5,3 milhões de americanos idosos vivos em 2017, ou aproximadamente 1 em cada 10 americanos idosos (https://www.alz.org/facts/) e inúmeros outros com demência. Para ajudar na luta para entender o que está afligindo nossos idosos, este código irá equipar os futuros pesquisadores e curiosos com a capacidade de monitorar o volume do cérebro ao longo do tempo.
Etapa 1: usando o Brain Box
Para usar a caixa de cérebro, é necessário apenas o seguinte:
- Varreduras de ressonância magnética de um cérebro e o nome e formato de tais arquivos (todos devem ter aproximadamente as mesmas dimensões)
- Comprimento de uma varredura
- Distância entre cada camada (varredura de ressonância magnética)
- Nome do paciente (ao inserir, não inclua espaços e coloque o nome e o sobrenome em maiúscula, como: NomeSobrenome)
E, a partir disso, é possível acompanhar as tendências de volume cerebral de um indivíduo ao longo do tempo. Assim, os números das tendências de Alzheimer podem ser rastreados por este software. O comprimento que usamos no teste foi de 180 mm para o comprimento de uma varredura e 5 mm para a distância entre as varreduras de ressonância magnética, com base em números médios.
No entanto, a aplicação da caixa do cérebro não precisa ser restrita a esta tarefa. Se as seções transversais de um determinado sólido são fotografadas, como um tumor em si, as tendências nas mudanças de volume para eles também podem ser rastreadas no software.
Etapa 2: Introdução: Analisando as seções transversais
Em estruturas tridimensionais, os planos bidimensionais em que consistem seriam chamados de seções transversais. Imagine que uma pilha de papéis forma um prisma retangular, então cada pedaço de papel seria uma seção transversal do papel. Ao imaginar o cérebro, aplicamos a mesma linha de pensamento. MRI (imagem por ressonância magnética) (ver informações sobre MRI) captura as seções transversais do cérebro e, usando os limites definidos em cada "camada" do cérebro fornecida, podemos construir uma estrutura para modelar e encontrar o volume do cérebro. No entanto, devemos primeiro construir uma função para fornecer informações sobre esses limites.
Etapa 3: Configurando uma função: LevelCurveTracings.m
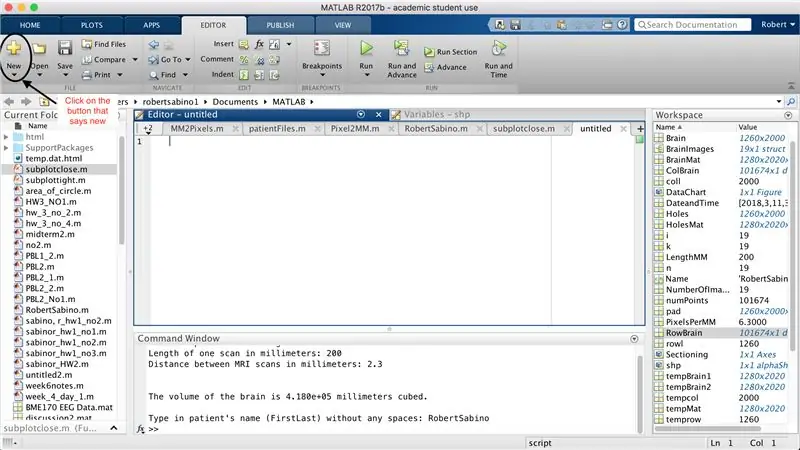
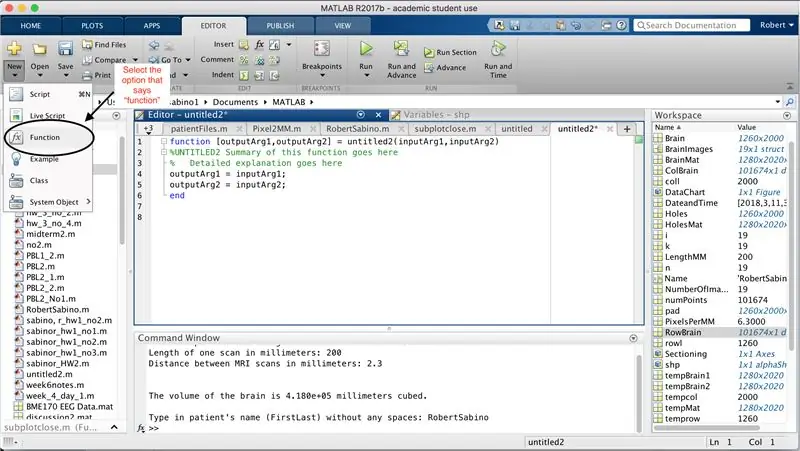
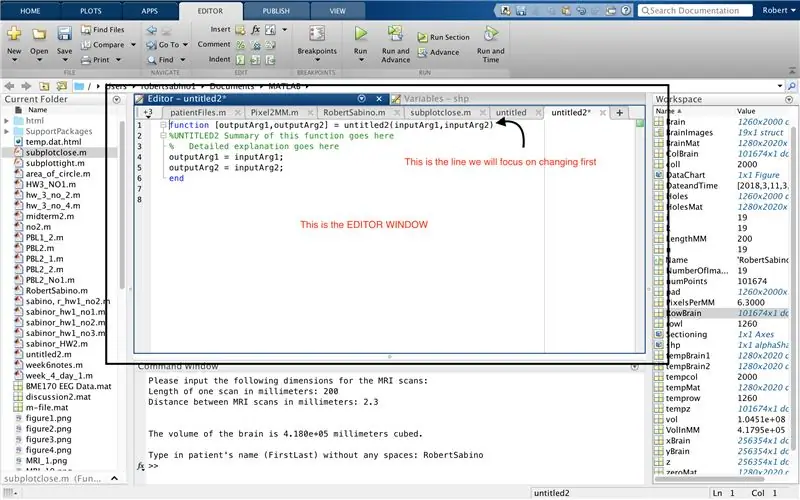

Primeiro, certifique-se de que o MATLAB_R2017b tenha sido baixado em seu computador (baixe aqui) e abra o MATLAB. Na interface do MATLAB, clique no botão no canto superior esquerdo da janela que diz "Novo" com um sinal de mais amarelo em negrito e selecione a opção "função", para abrir na janela do editor um espaço que se assemelha ao do terceira foto. Vamos nos concentrar em alterar a primeira linha para configurar a função. Onde diz "outputArg1", substitua por "brain", "outputArg2" para dizer "holes", "untitled2" para "exp2" e "inputArg1" para "image" e exclua "inputArg2". Agora você tem uma função a ser chamada usando "exp2", pegando um argumento "imagem" e gerando os limites de "cérebro" e "buracos". A primeira linha da função deve ser semelhante à linha representada na quarta imagem. Exclua todo o código abaixo desta linha inicial.
Etapa 4: desenvolvendo o algoritmo de limite: encontrando limites

Digite o código da seguinte maneira, abaixo da linha. Esta seção da função faz o seguinte linha por linha.
- Carregue a imagem "image" na variável "mri".
- Transforme "mri" em uma imagem feita de valores em uma faixa de números a uns e zeros (também conhecida como binarização) com base em um valor limite definido. Se o valor em um pixel for igual ou maior que 0,1, ele é definido como um; caso contrário, o valor naquele pixel é definido como zero.
- As quatro linhas a seguir transformam 10 colunas e linhas nas bordas da camada de varredura de MRI em zeros, para evitar a leitura de valores impróprios como limites (conforme aprendido com a experiência com o código).
- Na linha final, bwboundaries traça os limites da imagem binarizada "mri" e a define igual a "b", uma matriz com os elementos cujos índices correspondem aos dos limites definidos como um.
Etapa 5: Desenvolvendo o Algoritmo Bound: Gerando Matriz de Limite Externo
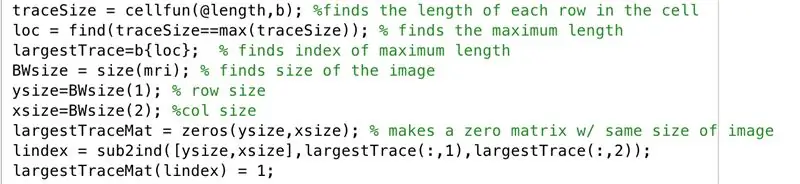
Acompanhe na janela do editor o seguinte código na imagem. Esta seção do código faz o seguinte linha por linha.
- Encontre o comprimento de cada uma das linhas da imagem binarizada "b" (cellfun aplica o comprimento da função a cada linha).
- Defina "loc" para armazenar os comprimentos máximos.
- Encontre o índice do comprimento máximo, definido para armazenar em "maiorTrace".
- Encontre o tamanho da imagem "mri", que consiste no mesmo tamanho que "b", e defina como "BWsize".
- Encontre o número de linhas na matriz da imagem, defina como "ysize".
- Encontre o número de colunas na matriz da imagem, defina como "xsize".
- Gere o array "maiorTraceMat", uma matriz "ysize" por "xsize" de zeros.
- Encontre o índice equivalente a partir dos valores subscritos correspondentes a onde estavam os maiores valores de Rastreio xey, armazene no vetor "lindex".
- Na matriz de zeros, "maiorTraceMat", transforme os elementos nos índices que correspondem aos valores de índice armazenados como elementos em "lindex" em uns.
Assim, a matriz lógica "maiorTraceMat" tem a maior região limitada da seção transversal de varredura do cérebro dada plotada como uns com um fundo de zeros
Etapa 6: Desenvolvendo o Algoritmo Bound: Trabalhando com o Ponto Central

Em seguida, devemos testar para ver se a seção transversal consiste em mais de uma região (a maior). Ao testar o alinhamento do centroide da maior região, podemos ver se há uma região contígua, o que produziria um centróide mais centrado, ou a possibilidade de várias regiões.
- Use "regionProps" para encontrar informações sobre os centróides presentes, definido igual ao array da estrutura "tempStruct"
- Matriz de formulário "centroids" com dados do campo "centroid" concatenados verticalmente
- Pegue os valores da segunda coluna de "centróides" (as coordenadas da dimensão horizontal)
- Execute um filtro para verificar o alinhamento do centróide com o centro horizontal
Etapa 7: Desenvolvendo o algoritmo vinculado: quando um centróide não está centrado
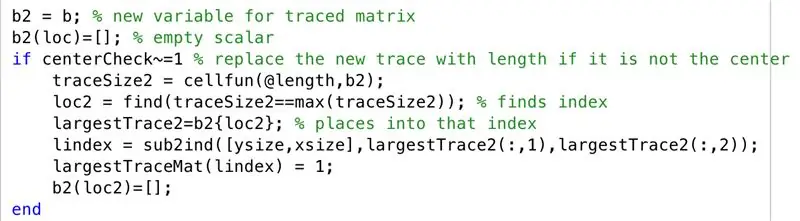
No cenário em que o centróide da maior região de rastreamento não está centralizado, passamos pelas etapas a seguir. Como observamos nas imagens de ressonância magnética, a tendência era ter hemisférios do cérebro retratados na seção transversal quando não contíguos, então agora continuamos a traçar o segundo maior traço junto com o maior traço no "maiorTraceMat"
- Defina a matriz rastreada para uma nova variável "b2"
- Inicialize a matriz vazia "b2", com um conjunto indexado por "loc"
- Crie uma condicional, para quando um centróide não estiver centralizado (ou seja, uma camada de várias regiões)
- Defina um novo tamanho de rastreamento a ser encontrado para cada linha (traceSize2)
- Defina "loc2" para encontrar os índices onde os limites estão presentes
- Deixe as células especificadas por "loc2" em "b2" serem iguais a "maiorTrace2"
- Converta os subscritos em índices, defina como "lindex"
- Altere os elementos correspondentes a "lindex" em "maioresTraceMat" para 1
- Inicialize a matriz vazia "b2", com um conjunto indexado por "loc2"
Etapa 8: Desenvolvendo o Algoritmo Bound: Interferência de Orifícios

Ao lidar com buracos, os valores armazenados em "b2" rastreiam estruturas diferentes do traço maior, e plotá-los em uma forma preenchida de "maiorTraceMat" revelará onde há buracos nas regiões do cérebro.
- Crie a matriz "illedMat ", que é uma forma preenchida de" maiorTraceMat"
- Crie a matriz "interferMat", uma matriz "ysize" por "xsize" de zeros
- Crie a matriz "interferenceloc", para armazenar os valores de "b2", concatenados verticalmente
- Crie a matriz "lindex" para armazenar os índices que correspondem a "interferenceloc"
- Para índices em "interferMat" que correspondem a "lindex", defina o valor como 1, criando uma região limitada diferente
Etapa 9: desenvolvendo o algoritmo de limite: localizando buracos, finalizando limites do cérebro e limites de buracos

- Defina a matriz "tempMat" igual a "interferMat" mais "PreenchidoMat", adicionando assim cada valor na matriz uns aos outros
- Defina a matriz "holesLoc" igual aos índices onde "interferMat" e "Preenchido" ambos eram iguais a um
- Configure "holesMat" como uma matriz zero de dimensões "ysize" x "xsize"
- Defina índices em "holesMat" que são iguais a "holesLoc" como uns
- Defina "cérebro" como "maiorTraceMat"
- Defina "holes" como "holesMat"
Com a descoberta de onde os valores das matrizes adicionadas eram iguais a 2, os locais dos furos foram facilmente protegidos e plotados em uma matriz vazia.
Etapa 10: Registro de dados: Function PatientFiles.m
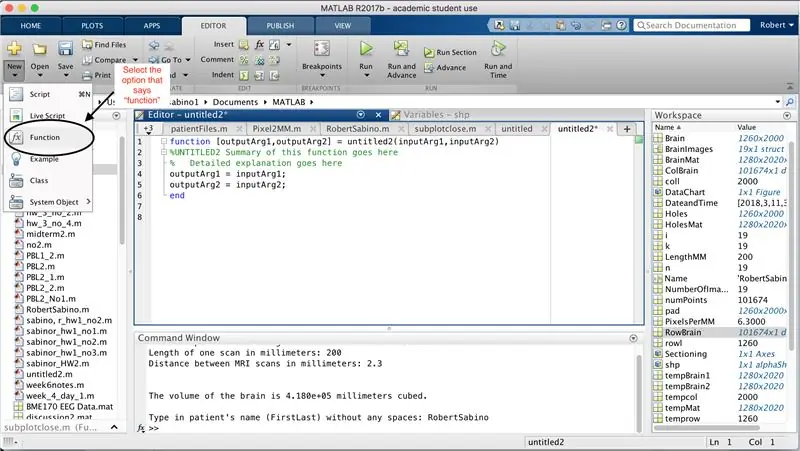
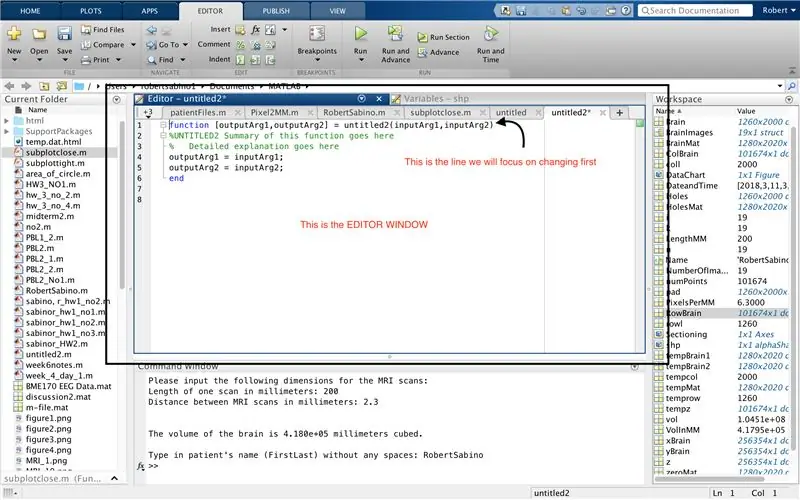

Muito parecido com a configuração da última função, clique no botão no canto superior esquerdo da janela que diz "Novo" com um sinal de adição amarelo em negrito e selecione a opção "função", para abrir na janela do editor um espaço que assemelha-se ao da terceira foto. Na primeira linha, exclua a matriz de saída e substitua apenas por "saída", substitua "untitled2" por "patientFiles", exclua todos os argumentos de entrada e siga a formatação especificada na quarta imagem da linha de código. A primeira linha desta função deve corresponder à formatação da imagem.
Etapa 11: registro de dados em arquivos
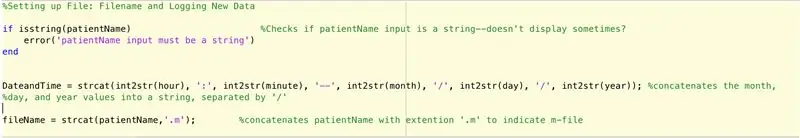

Para configurar um arquivo para registrar os dados encontrados pela função principal (ainda a serem descritos), devemos seguir estes passos (conforme prescrito pelo código linha a linha).
- Verifique se a entrada para nomedopaciente é uma string.
- Se não for uma string, exiba que a entrada do nomedopaciente deve ser uma string.
- Finalize a instrução if (evita erros).
- Configure uma declaração de string "Data e Hora" que fornecerá o seguinte formato: hora: minutos - mês / dia / ano.
- Defina a variável fileName como o seguinte: patientName.m.
Agora, para a próxima seção da função: Já existe um arquivo com este nome?
1) Suponha que o arquivo com este nome já exista:
- Execute o arquivo para obter os valores do passado enfileirados
- Adicione os dados "DateandTime" da iteração atual como uma nova célula na matriz de células de valores x (índice final + 1)
- Adicione o valor "brainVolume" atual como uma nova célula na matriz de células de valores y (índice final + 1)
- Salve as variáveis atuais carregadas no arquivo.
2) Suponha que o arquivo com este nome não exista:
- Crie um novo arquivo com o nome armazenado na variável "nomedopaciente"
- Adicione os dados "DateandTime" atuais como uma célula na matriz de células vazia de valores x
- Adicione os dados "brainVolume" atuais como uma célula na matriz de células vazia de valores y
- Salve as variáveis atuais carregadas no arquivo.
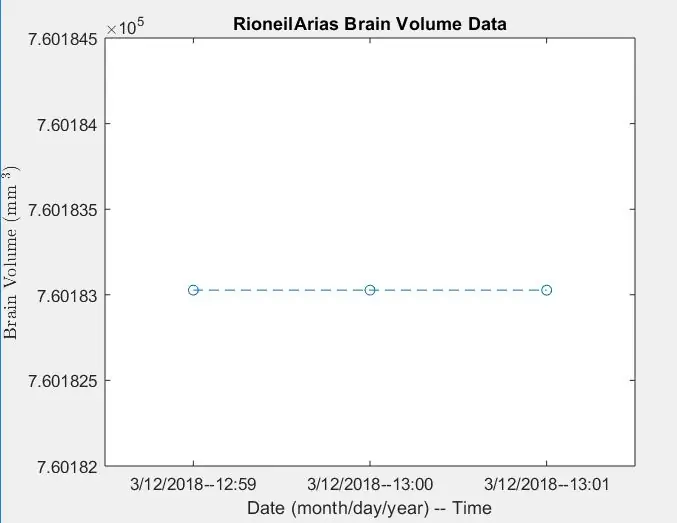
Etapa 12: Registro de dados: exibição de um gráfico do volume do cérebro ao longo do tempo

- Converta a matriz de valores x (xVals) em uma matriz categórica (xValsCategorical), para permitir a plotagem
- Gerar janela de figura 5
- Trace os pontos designados por "xValsCategorical" e "yVals" (contendo o volume do cérebro), usando círculos vazios para indicar os pontos e serem conectados por linhas tracejadas
- Título do gráfico como: PatientName Brain Volume Data
- Rotule o eixo x como mostrado na imagem
- Rotule o eixo y como mostrado na imagem
- Deixe a figura 5 igual à saída
A partir disso, a função patientName que está sendo chamada produzirá um arquivo com os dados editados, acompanhando o volume do cérebro ao longo do tempo e um gráfico exibindo as tendências.
Etapa 13: Fechando lacunas em subtramas: Subplotclose.m

A função, adaptada do código de https://www.briandalessandro.com, funciona para fechar as lacunas entre as figuras da subtrama do código principal, quando as figuras exibindo as imagens de ressonância magnética e as camadas do cérebro são criadas. A função subplot usada em subplotclose.m ajusta a posição dos subplots fornecidos para se ajustarem confortavelmente uns aos outros no aspecto da dimensão mais longa. Por exemplo, se o código pretende uma matriz 7 x 3, as linhas se ajustam perfeitamente, pois a dimensão da linha é mais longa. Se o código pretende uma matriz 3 x 7, as colunas se encaixarão bem, com lacunas nas linhas, conforme mostrado nas figuras de nosso código principal.
Etapa 14: O código principal: limpar tudo e solicitar entradas

Para iniciar o código principal, clique no mesmo botão que diz "Novo" no canto superior esquerdo da janela e selecione "Script" em vez de "Função" nas seções anteriores. Digite o código conforme mostrado na imagem dentro da janela do editor. As linhas de código realizam as seguintes tarefas em ordem:
- Feche todos os arquivos abertos, exceto 0, 1 e 2.
- Feche todas as janelas da figura.
- Limpe todas as variáveis na área de trabalho.
- Limpe a janela de comando.
- Exibir na janela de comando: Insira as seguintes dimensões para os exames de ressonância magnética:
- Em uma nova linha na janela de comando, pergunte: Comprimento de uma varredura em milímetros:. A resposta colocada pelo usuário será configurada para a variável "lengthMM".
- Em uma nova linha, pergunte: Distância entre as varreduras de ressonância magnética em milímetros:. A resposta colocada pelo usuário será definida para a variável "ZStacks".
Etapa 15: O código principal: processamento em lote das imagens

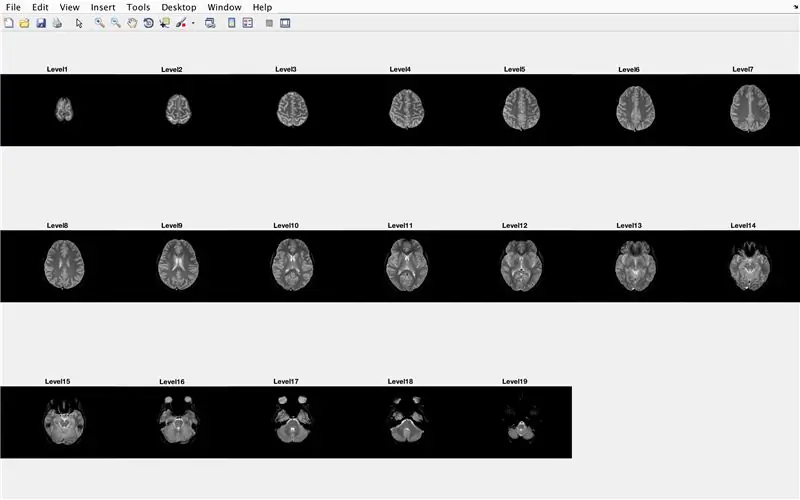
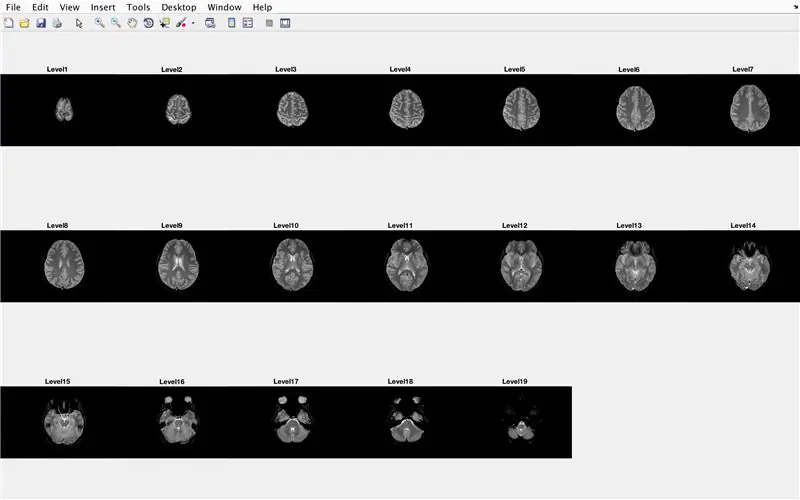
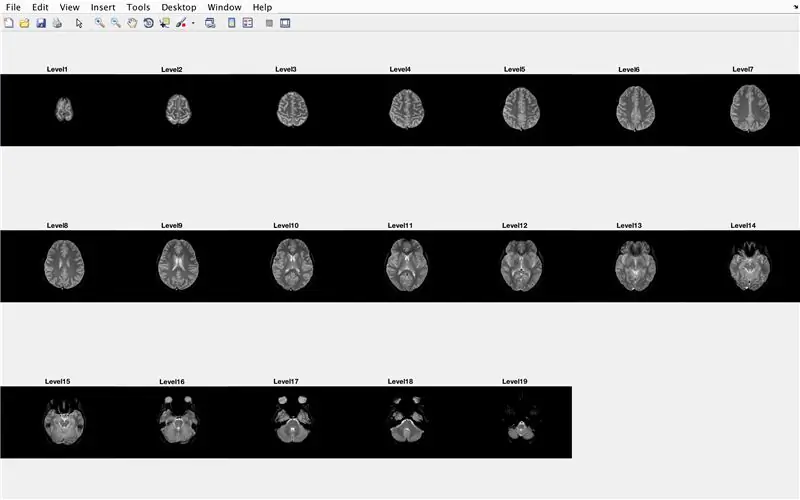
Nesta seção, o código carregará as imagens (consistindo em varreduras de ressonância magnética das seções transversais do cérebro) e armazenará os nomes de cada arquivo de imagem na variável "Base" e exibirá cada uma das varreduras de ressonância magnética. Siga com o código na imagem, que faz o seguinte:
- Crie um array de estrutura "BrainImages" que contém informações sobre todos os arquivos na pasta atual que se encaixam no formato de nome de MRI _ (). Png
- Defina a variável "NumberofImages" igual ao número de elementos na estrutura da matriz "BrainImages"
- Abra a janela da figura 1
- Defina um loop for para percorrer o número de imagens contadas no arquivo
- Para cada loop, "CurrentImage" é o respectivo nome de cada arquivo MRI_i.png, com o número da iteração como 'i'
- Gere uma subtrama 3 x 7 para exibir as 19 imagens a serem carregadas por "imshow"
- Exibir cada imagem como outro elemento na janela da subtrama
- Dê um título a cada elemento do subplot como Level_, onde em branco é o número da iteração do loop for.
- Fim do loop for (evitando erros)
Isso exibirá na janela da figura 1 todos os exames de ressonância magnética em formato bruto em uma configuração 3 x 7 sem lacunas na orientação x.
Etapa 16: O Código Principal: Preenchimento

Com o preenchimento, evitamos o problema de pequenas discrepâncias nos tamanhos da imagem que podem gerar erros de incompatibilidade de dimensão no caso de uma imagem ser ligeiramente maior que a outra.
- Abra a janela da figura 2
- Carregue a matriz de imagem de MRI_1-p.webp" />
- Encontre o tamanho da matriz da imagem e defina como "OriginalXPixels" (para o número de linhas) e "OriginalYPixels" (para o número de colunas)
- Configure a matriz "BrainMat" para consistir em todos os zeros com mais 20 linhas e mais 20 colunas para cada plano e 19 seções transversais totais, uma por plano.
- Configure "HolesMat" para consistir na mesma matriz tridimensional de zeros para inserir as coordenadas do furo mais tarde
- Crie "zeroMat" com o tamanho de um bloco mais vinte linhas e vinte colunas, uma matriz bidimensional de zeros.
Etapa 17: O Código Principal: Determinando Limites
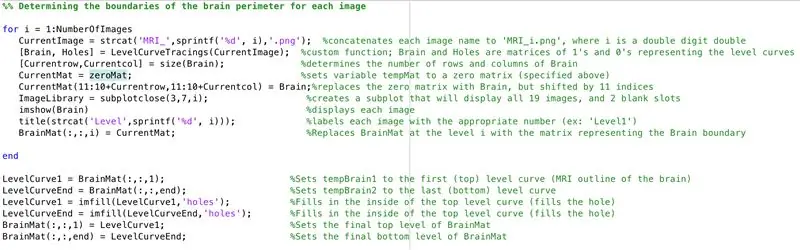
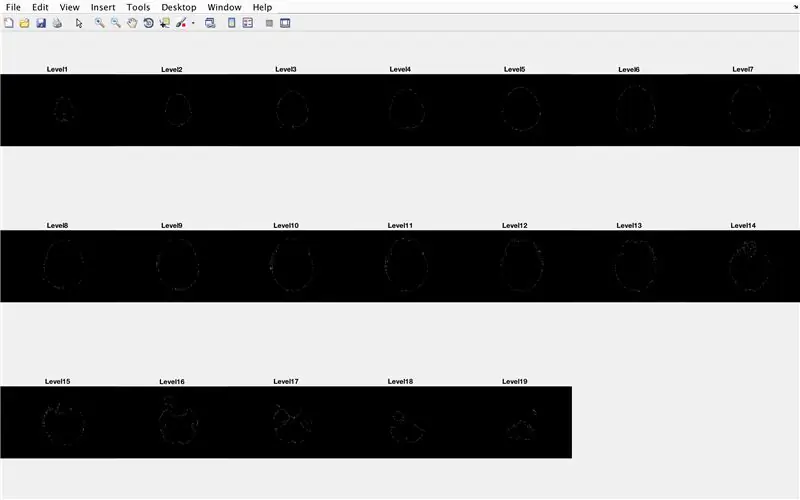
- Defina um loop for para percorrer os dados de cada imagem carregada anteriormente
- Da mesma maneira do processamento em lote anterior, "CurrentImage" carrega arquivos com "MRI_i.png", onde i é o número da iteração
- Execute cada imagem por meio da função de processamento "LevelCurveTracings2.m" que você criou anteriormente
- Encontre o tamanho da saída "Brain", defina o número de linhas como "Currentrow" e o número de colunas como "Currentcolumns"
- Defina "CurrentMat" para uma matriz de zeros com as dimensões especificadas por "Currentrow" e "Currentcolumns"
- Centralize os dados de "Brain" em "CurrentMat", com uma margem de 10 linhas em todos os lados
- Gere um subplot de dimensões 3 x 7, para exibir os limites das imagens
- Dê um título a cada um dos elementos do subplot na janela da figura
- Gerar matriz tridimensional "BrainMat" composta de cada camada de limites "CurrentMat"
- Finalize o loop for (para evitar erros)
A subseção a seguir preenche os orifícios deixados na parte superior e inferior da forma tridimensional proposta
- Defina "LevelCurve1" igual à primeira camada de "BrainMat" (parte inferior do sólido)
- Defina "LevelCurveEnd" igual à camada final de "BrainMat" (topo do sólido)
- Substitua "LevelCurve1" por uma camada preenchida
- Substitua "LevelCurveEnd" por uma camada preenchida
- Defina a camada preenchida como a camada inferior de "BrainMat"
- Defina a camada preenchida como a camada superior de "BrainMat"
Etapa 18: O Código Principal: Determinando a Dimensão Z Adequada

As primeiras três linhas consistem em configurar um array vazio "z" e fazer operações de conversão simples (dividir os pixels pelo comprimento) para obter uma leitura adequada do volume em mm ^ 3
- Crie um loop for para percorrer cada camada
- Encontre o número de unidades em uma determinada camada
- Converta as coordenadas z para aqueles em valores que são escalados para a proporção adequada, defina como "tempz", um vetor de coluna
- Adicione o valor z para a curva de nível ao vetor z
Com isso, as coordenadas z são ajustadas corretamente.
Etapa 19: O Código Principal: Determinando as Coordenadas X e Y

Agora, para determinar as posições xey de cada um dos pontos nos limites.
- Inicialize "xBrain" como uma matriz vazia
- Inicialize "yBrain" como uma matriz vazia
- Configure um loop for para percorrer cada imagem carregada
- Compile uma matriz de duas colunas para armazenar as coordenadas planas de cada ponto no limite, representadas pelos vetores de coluna "RowBrain" e "ColumnBrain"
- Anexe "xBrain" com as coordenadas "RowBrain" atualmente encontradas
- Anexe "yBrain" com as coordenadas "ColumnBrain" atualmente encontradas
- Finalize o loop for (para evitar erros)
Etapa 20: O código principal: traçando uma estrutura tridimensional, encontrando o volume e registrando os dados
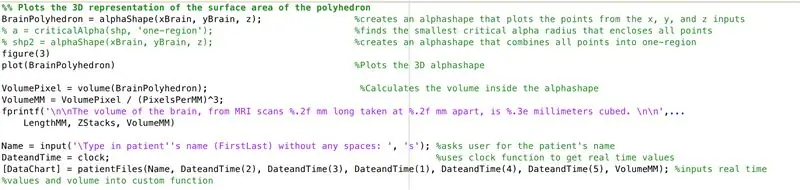

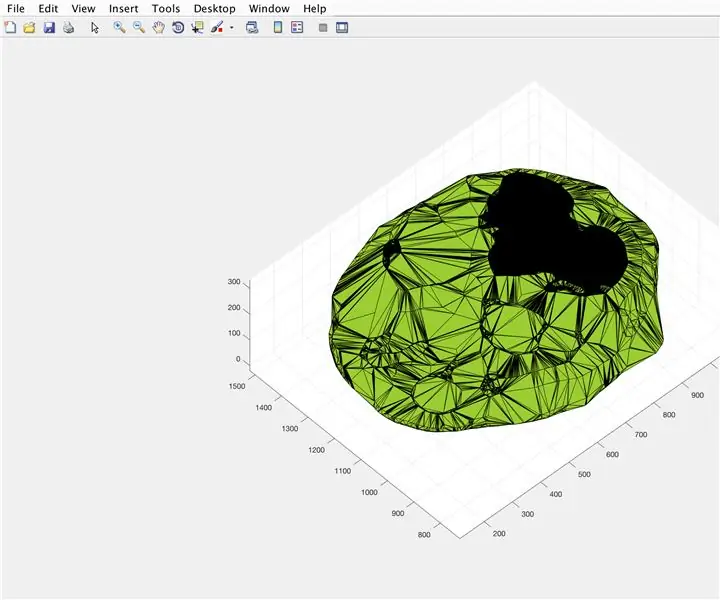
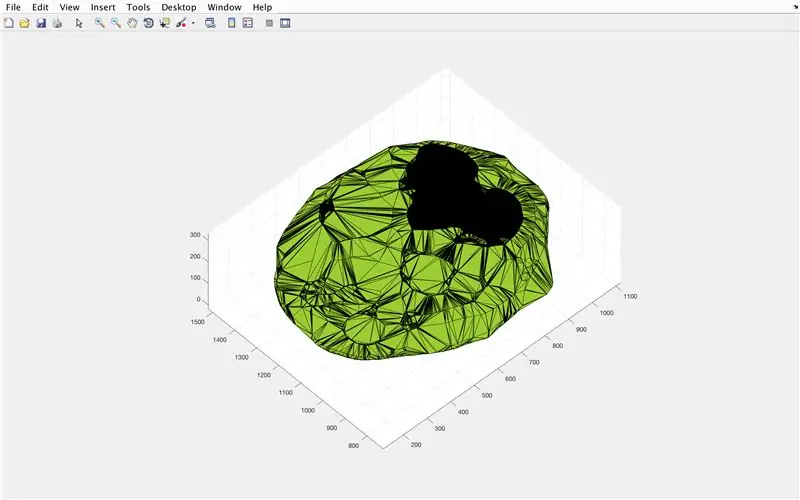
Usando a função alphaShape, criaremos uma estrutura tridimensional a partir da qual podemos calcular o volume do cérebro.
- Use a função alphaShape, insira os vetores "xBrain", "yBrain" e "z" para as coordenadas x, y e z e defina igual a "BrainPolyhedron"
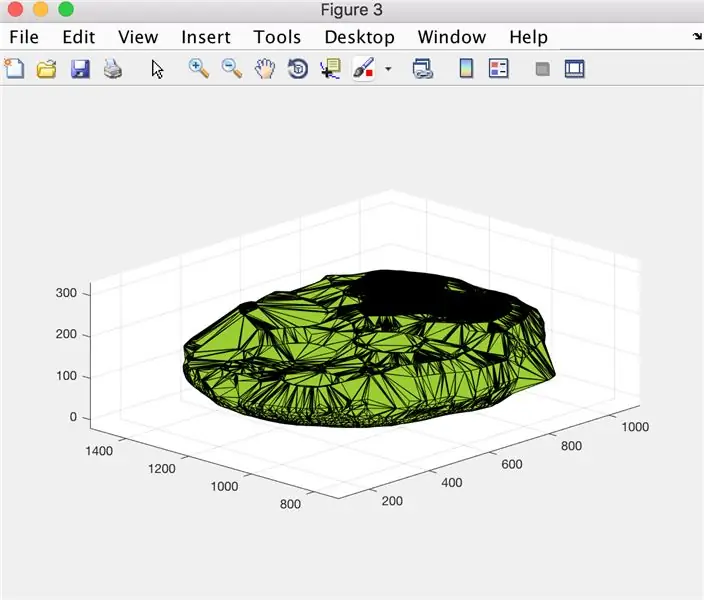
- Abra a janela da figura 3
- Trace a forma alfa calculada "BrainPolyhedron", exibida na janela da figura
- Calcule o volume da forma alfa, usando uma função "volume" que funciona para formas alfa
- Converta o volume em mm ^ 3
- Imprime o volume do sólido na janela de comando
- Solicita que o nome de um paciente seja definido como uma entrada
- Obtenha a data e a hora atuais com o relógio e defina como "Data e Hora"
- Chame a função "patientFiles" para registrar e plotar os dados calculados
A partir daqui, a segunda e a terceira imagens devem mostrar as figuras que aparecem, e a quarta imagem aquela que deve ser exibida na janela de comando.
Recomendado:
Introdução aos sensores de vibração e temperatura sem fio de longo alcance: 7 etapas

Introdução aos sensores de vibração e temperatura sem fio de longo alcance: Às vezes, a vibração é a causa de problemas sérios em muitas aplicações. De eixos e rolamentos da máquina ao desempenho do disco rígido, a vibração causa danos à máquina, substituição antecipada, baixo desempenho e inflige um grande impacto na precisão. Monitorando
Registrador de ECG - um monitor cardíaco vestível para aquisição e análise de dados de longo prazo: 3 etapas

ECG Logger - um monitor cardíaco vestível para aquisição e análise de dados de longo prazo: Primeira versão: outubro de 2017 Última versão: 1.6.0Status: StableDifficulty: HighPrerequisite: Arduino, Programming, Hardware buildingUnique repository: SF (veja os links abaixo) Suporte: Somente fórum, nenhum registrador PMECG é um monitor cardíaco vestível por muito tempo
Acompanhamento de variações de aceleração com Raspberry Pi e MMA7455 usando Python: 6 etapas

Rastreando variações de aceleração com Raspberry Pi e MMA7455 usando Python: Eu não tropecei, estava testando a gravidade. Ainda funciona … A representação de um ônibus espacial em aceleração esclareceu que um relógio no ponto mais alto do ônibus pegaria mais rápido do que um na base por causa da expansão do tempo gravitacional. Algum
Exibição de tempo e tempo do Arduino 3 em 1: 11 etapas

Arduino 3 em 1 Time and Weather Display: Eu gosto de microcontroladores PIC e gosto de programar em linguagem assembly. Na verdade, nos últimos dois anos, postei cerca de 40 projetos em meu site com base nessa combinação. Recentemente, eu estava encomendando algumas peças de um dos meus favoritos dos EUA v
Como trocar a bateria em uma etiqueta de tempo de tempo: 12 etapas

Como trocar a bateria em uma etiqueta de tempo Tempo: As etiquetas de tempo de tempo do navio Vessel são um substituto legal para um relógio, prendendo em roupas, alças de bolsa ou bordas de bolso. A bateria acaba eventualmente, então veja como substituí-la. É uma bateria 364 / AG1 / LR621 / SR621W / 164 padrão que
