Índice:
- Autor John Day day@howwhatproduce.com.
- Public 2024-01-30 11:35.
- Última modificação 2025-01-23 15:03.
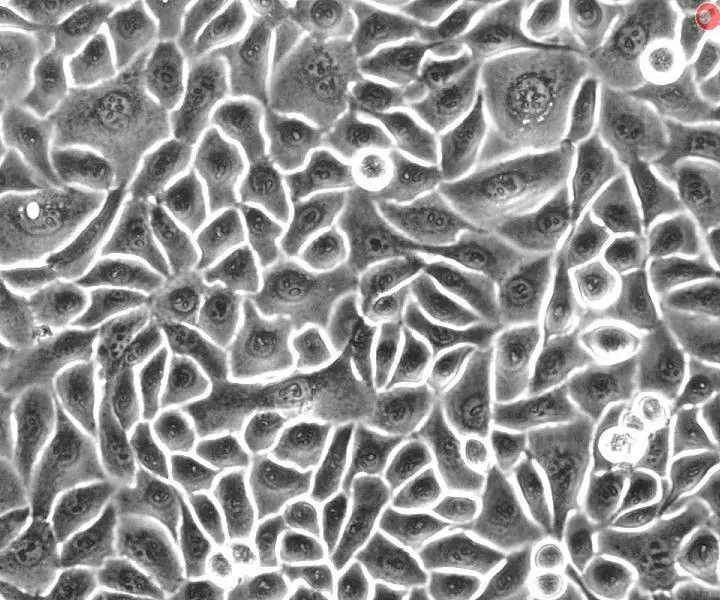
Nosso projeto Sandbox tem como objetivo ajudar os pesquisadores no campo biológico a analisar amostras de células e descobrir as condições de suas células. Depois que o usuário insere uma imagem de sua amostra de célula, nosso código processa a imagem para deixá-la pronta para a contagem de células, convertendo a imagem em tons de cinza e binário. O código utiliza o limiar para remover o excesso de informações não pertencentes às células reais, a fim de encontrar com precisão as massas celulares. Depois de contar o número de células na imagem, nosso código fornece o tamanho do pixel da imagem e, em seguida, encontra a distribuição da área da célula traçando o número de células versus a área de células para dar ao usuário a confluência, que é o porcentagem da superfície de uma placa de cultura que é coberta pelas células de interesse. Com base na confluência, o pesquisador pode determinar se as células devem ou não ser passadas; A passagem de células refere-se à expansão da vida ou do número de células ou microrganismos, transferindo parte da cultura para um meio de crescimento fresco, e é útil quando as células crescem muito ou ficam sem comida. Depois de executar o código e processar a imagem, o usuário pode decidir se aceita ou rejeitar os resultados e continuar a ajustar o limite para melhor recuperação dos dados, se necessário.
Etapa 1: Selecione e configure a imagem para análise
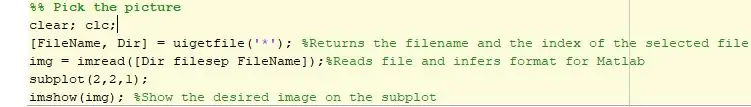
O primeiro passo em nosso código é selecionar a imagem apropriada e configurá-la para análise em Matlab. Podemos selecionar a imagem usando a função uigetfile que nos permite pegar qualquer arquivo e inseri-lo no programa. Então, usando o imread lemos a imagem e a configuramos para análise no Matlab. A imagem selecionada é exibida em uma subtrama.
Etapa 2: Limiar e GUI
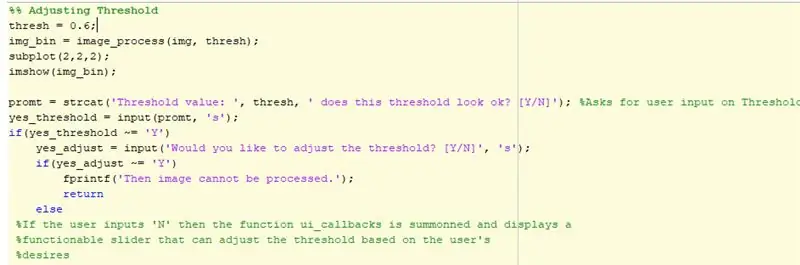
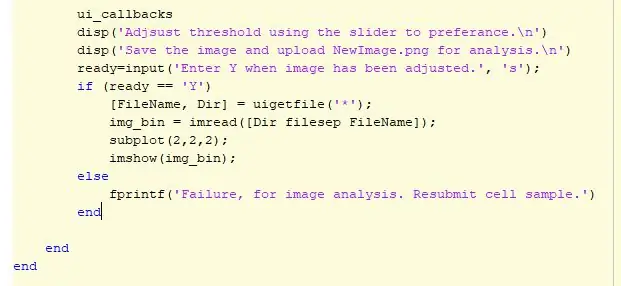
No início do código, uma imagem é escolhida usando “uigetfile” e, em seguida, definindo essa imagem com uma variável. A variável será então usada para identificar o código ao executar análises diferentes. Uma subtrama 2x2 é criada na figura. Na posição 1, a imagem original será exibida. A próxima seção do código é onde ocorrem os ajustes dos limites. Inicialmente, um padrão de um limite de 0,6 é usado e exibido na posição 2 da subtrama. Uma instrução if é então usada para determinar se o usuário deseja manter o limite ou ajustá-lo. O usuário pode então ajustar o limite usando uma GUI que inclui a imagem em diferentes condições de limite, o controle deslizante e um botão Salvar. Depois de definir o limite, o usuário clicará no botão salvar para salvar a imagem e ela será armazenada nos arquivos MATLAB do usuário como um-p.webp
Etapa 3: traçar contornos e distribuição de células
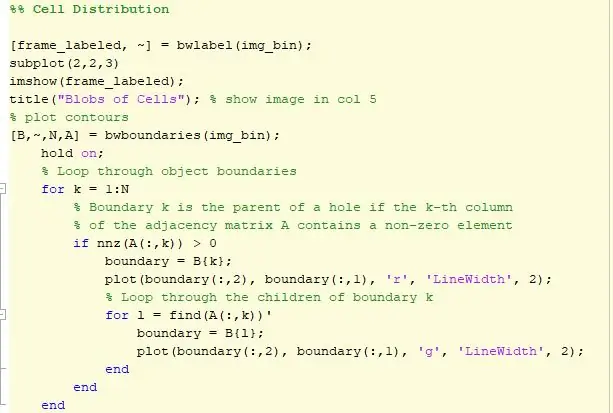
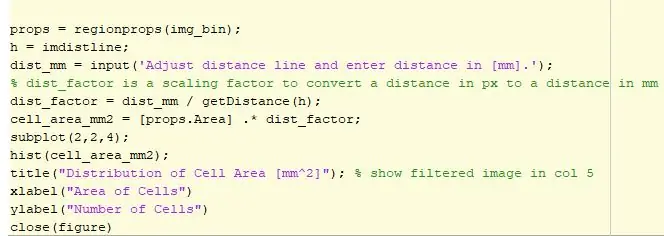
A próxima parte do código plota os contornos. Há um loop no qual as células são envolvidas por um perímetro vermelho e as células que estão no topo de outra célula são destacadas em verde. A imagem delineada é então exibida na posição 3 com uma linha de distância interativa. Esta linha determinará o número de pixels na linha ajustados pelo usuário para um conversor de pixel em milímetro. O fator de distância é então multiplicado pela área determinada pelo regionprops e a área agora é expressa em milímetros ao quadrado. Os dados são então plotados usando um histograma para ver a distribuição das células por sua área. Este histograma será então exibido na posição 4.
Etapa 4: converter a imagem da célula

Nesta etapa, pegamos a imagem em tons de cinza e a binarizamos, filtramos e invertemos. A execução dessas funções na imagem removeu pixels barulhentos que poderiam ser confundidos com células e tornou a imagem mais suave e suave ao redor das bordas das células. Isso foi feito para extrair as células na imagem como "bolhas" distinguíveis que diferiam em intensidade do fundo. Os "Blobs" eram imagens brancas de alta intensidade e o fundo era preto. Se fosse permitido um pouco mais de tempo, teríamos usado uma conversão de imagem Blob diferente em vez da função imbinarizar para ser mais preciso e mais adequado às nossas imagens, mas precisávamos de mais tempo para pesquisar e implementar a função.
Etapa 5: Contar células e calcular a confluência celular

Nesta etapa do código, pretendemos contar o número de células que estavam na imagem. Usamos principalmente a função regionprops para calcular as áreas dos blobs e se a área foi calculada para estar em nosso limite desejado, ela seria plotada em um subplot. Os limites foram definidos para remover pequenos pixels com ruído ou grandes intensidades que não eram células. O contador de células então contaria os centróides que foram plotados e os adicionaria ao contador no loop for. Uma vez que as áreas das células foram determinadas, pudemos calcular a confluência. A principal importância desta etapa no código era encontrar a confluência das células e isso foi fundamental para o nosso objetivo final do código. Fizemos isso calculando somando os pixels (sum (allAreas)) em cada blob e depois dividindo pelo valor total de pixels da imagem (numel (img)). Essa proporção nos daria a confluência e se fosse determinada como maior que 80% então é hora de as células serem passadas pelo pesquisador. Nosso objetivo era ser o mais preciso e preciso possível, mas com o tempo limitado para que ocorresse imprecisão. Se o tempo permitisse, teríamos procurado maneiras de tornar a contagem de bolhas mais precisa, como mais técnicas de filtragem e / ou uma Transformada de Hough, pois ainda não foi feita pesquisa suficiente para experimentar essa técnica de filtragem
Etapa 6: Redondeza das células
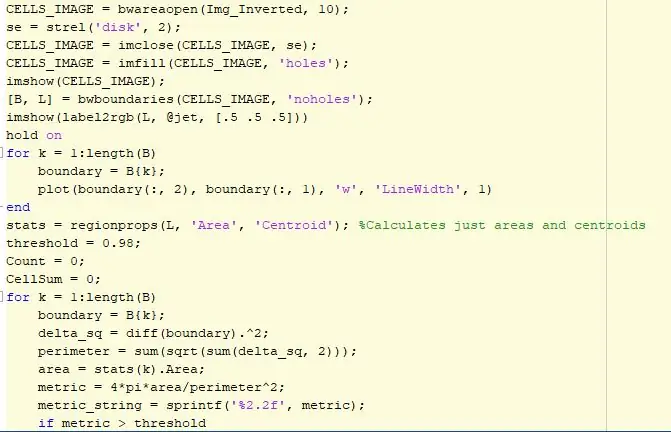

Antes de poder medir a redondeza do blob em uma imagem, precisamos converter RGB para tons de cinza, binarizar, inverter e filtrar a imagem. Uma técnica de filtragem está usando a função bwareaopen, que filtra a imagem de interesse e remove quaisquer especificações ou pixels que são muito pequenos que não representam o tamanho de uma célula. Um elemento estruturante é criado com a forma de um disco e vizinhança de 2 e é usado para preencher quaisquer lacunas no fundo ou dentro das células. Em seguida, usamos a função bwboundaries que rastreia os blobs e os armazena em uma matriz. Em seguida, rotulamos a imagem usando cores diferentes para que se torne um visual mais claro. Em seguida, usando um loop for executado de acordo com o número de objetos e buracos encontrados na imagem, ele define um limite em torno dos blobs correspondentes a esse array. Uma vez que este loop termina, outro loop começa, novamente de acordo com o número de objetos e buracos encontrados na imagem. Desta vez, usamos a função regionprops que reúne certas propriedades como área do array e armazena a informação contendo o número de objetos e buracos. Usando essas informações, calculamos a área e o perímetro desses objetos usando a forma do centróide. Um limite é definido para comparar os resultados, uma vez que calculamos a unidade métrica de um objeto circular e plota essa medida como texto ao lado dos centróides calculados. O código final exibirá o arredondamento das diferentes células encontradas na imagem e quaisquer valores próximos ao valor de um serão mais arredondados do que os outros. Um valor de 1 implica que a célula é perfeitamente redonda e está em boas condições para ser passada.
Recomendado:
Projeto do oscilador baseado no modo de corrente para amplificadores de potência de áudio Classe D: 6 etapas
Projeto do oscilador baseado no modo atual para amplificadores de potência de áudio Classe D: Nos últimos anos, os amplificadores de potência de áudio Classe D tornaram-se a solução preferida para sistemas de áudio portáteis, como MP3 e telefones celulares, devido à sua alta eficiência e baixo consumo de energia. O oscilador é uma parte importante da classe D au
ECG-BME 305 Automatizado Crédito Extra do Projeto Final: 7 Passos

ECG- BME 305 Automatizado Projeto Final Crédito Extra: Um eletrocardiograma (ECG ou EKG) é usado para medir os sinais elétricos produzidos por um coração batendo e desempenha um grande papel no diagnóstico e prognóstico de doenças cardiovasculares. Algumas das informações obtidas de um ECG incluem o ritmo
BME 305 EEG: 4 etapas

BME 305 EEG: um eletroencefalograma (EEG) é um dispositivo usado para medir a atividade elétrica do cérebro de um sujeito. Esses testes podem ser muito úteis no diagnóstico de diferentes distúrbios cerebrais. Ao tentar fazer um EEG, existem diferentes parâmetros que precisam
Projeto Sandbox: Cálculo e Interpretação BAC: 6 etapas

Projeto Sandbox: Cálculo e Interpretação de TAS: Por Haarika Gogineni, Hana Schlosser e Benedict UisecoNeste projeto, tentaremos calcular a Concentração de Álcool no Sangue (TAS) com base no número de bebidas, peso e gênero de um sujeito. Após a saída do BAC calculado, iremos declarar o
Gravador alimentado por USB! Este projeto pode queimar plásticos / madeira / papel (o projeto divertido também deve ser uma madeira muito fina): 3 etapas

Gravador alimentado por USB! Este projeto pode queimar através de plástico / madeira / papel (projeto divertido também deve ser madeira muito fina): NÃO FAÇA ISSO USANDO USB !!!! Eu descobri que isso pode danificar seu computador com todos os comentários. meu computador está bem tho. Use um carregador de telefone 600ma 5v. Eu usei isso e funciona bem e nada pode ser danificado se você usar um plugue de segurança para interromper a alimentação
