Índice:
- Autor John Day day@howwhatproduce.com.
- Public 2024-01-30 11:35.
- Última modificação 2025-01-23 15:03.
Por: Phuc Lam, Paul Yeung, Eric Reyes
Saber que erros na segmentação pulmonar produzirão informações falsas sobre a identificação de uma área de doença e podem afetar diretamente o processo de diagnóstico. As técnicas modernas de auxílio ao computador não conseguiram fornecer resultados precisos quando as doenças pulmonares têm formas desafiadoras. Essas formas anormais podem ser causadas por derrames pleurais, consolidações, etc. Aplicando a técnica de segmentação pulmonar, na qual os limites do pulmão são isolados do tecido torácico circundante, nosso aplicativo pode identificar os limites com os limites de entrada do usuário para fornecer visualizações totalmente personalizáveis das formas dos pulmões, O objetivo deste projeto MatLab é criar um aplicativo de segmentação pulmonar interativo e amigável para detectar condições patológicas nas imagens de raios-X dos pulmões. Nosso objetivo é criar uma forma mais eficaz de ilustrar e identificar pulmões anormais, a fim de fornecer aos médicos e radiologistas uma forma mais confiável de diagnosticar doenças pulmonares. Usando a ferramenta de criação de aplicativos no MatLab, o programa foi projetado para funcionar especificamente com exames de raio-x de tórax e tomografia computadorizada (TC), mas também foi testado para funcionar com exames de ressonância magnética.
As instruções abaixo contêm nossa técnica de filtragem de ruído (filtro passa-baixo de Wiener), bem como o limiar da imagem (usando o histograma de intensidade da imagem em tons de cinza) e usando um gradiente morfológico (a diferença entre a dilatação e a erosão de uma imagem) para identificar uma região de interesse. A instrução explicará como integramos todos os elementos na interface gráfica do usuário (GUI).
Observação:
1). Este projeto é inspirado por um artigo de pesquisa: "Segmentação e análise de imagem de pulmões anormais em TC: abordagens atuais, desafios e tendências futuras". Que pode ser encontrado aqui
2). Estamos usando imagens de raios-X do NIH: Clinical Center. O link pode ser encontrado aqui
3). A ajuda do designer de aplicativos pode ser encontrada aqui
4). Antes de executar o código: você precisa alterar o caminho Dir (na linha 34) para o diretório do seu arquivo e tipo de imagem (linha 35) (estamos analisando *.png).
Etapa 1: Etapa 1: Carregando a imagem
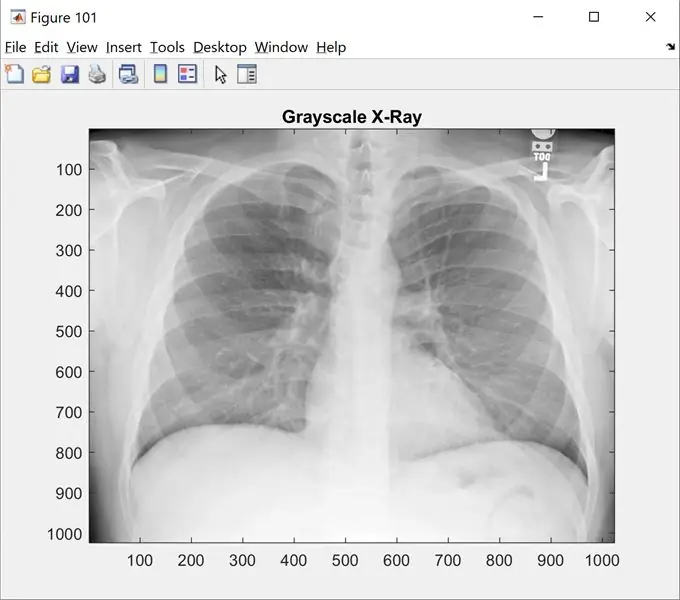
Esta etapa mostrará a imagem original em escala de cinza. Altere o 'name_of_picture.png' para o nome da sua imagem
Claro; clc; feche tudo;
%% Carregando imagens
raw_x_ray = 'name_of_picture.png';
I = imread (raw_x_ray);
figura (101);
imshow (I);
mapa de cores (cinza);
título ('Grayscale X-Ray');
Etapa 2: Etapa 2: Filtragem de ruído e histograma
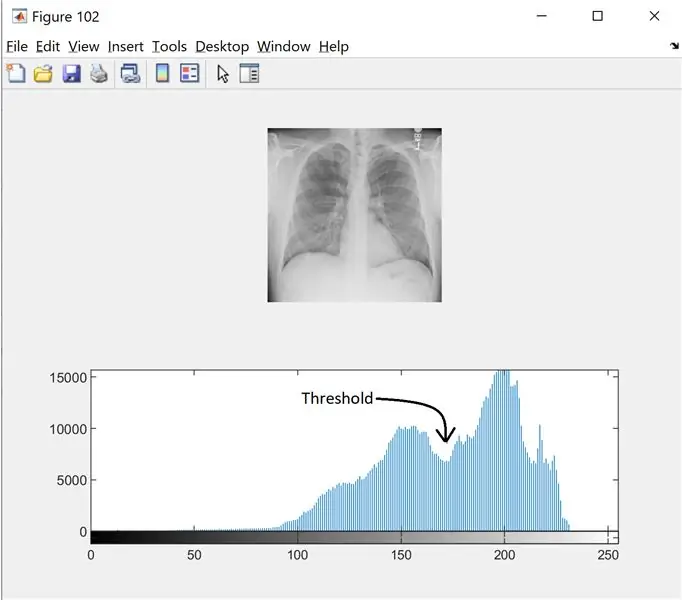
A fim de encontrar o limite para a imagem em escala de cinza, olhamos para o histograma para ver se há modos distintos. Leia mais aqui
I = wiener2 (I, [5 5]);
figura (102);
subtrama (2, 1, 1);
imshow (I);
subtrama (2, 1, 2);
imhist (I, 256);
Etapa 3: Etapa 3: Definir limites
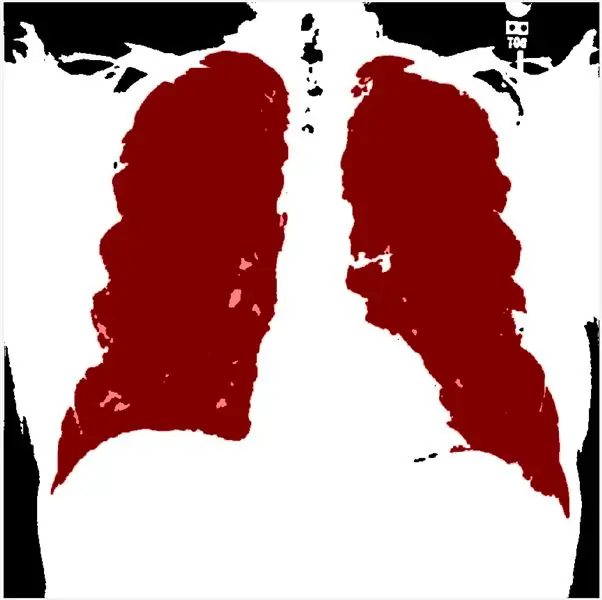
Esta etapa permite definir o limite de acordo com o histograma. morphologicalGradient destacará a região de interesse em vermelho e a função visboundaries sobrepõe a imagem delineada e filtrada do pulmão em vermelho.
Usando regionprops, podemos determinar as matrizes de solidez e classificá-las em ordem decrescente. Em seguida, binarizo a imagem de sclae cinza e aplico o método do gradiente morfológico e o mLoren Shurasking para destacar a região de interesse (ROI). A próxima etapa é inverter a imagem para que a ROI do pulmão fique branca no fundo preto. Eu uso a função showMaskAsOverlay para exibir 2 máscaras. Nota: o código é inspirado em Loren Shure, link.
Por último, crio um contorno vermelho usando os limites bwbw e mascarando a imagem de filtro e os limites.
a_thresh = I> = 172; % definiu este limite
[labelImage, numberOfBlobs] = bwlabel (a_thresh);
props = regionprops (a_thresh, 'all');
SortSolidity = sort ([props. Solidity], 'descer');
SB = solidez classificada (1);
se SB == 1% SB aceita apenas solidez == 1 filtra os ossos
binaryImage = imbinarize (I); figura (103);
imshow (binaryImage); mapa de cores (cinza);
SE = strel ('quadrado', 3);
morphologicalGradient = imsubtract (imdilate (binaryImage, SE), imerode (binaryImage, SE));
máscara = imbinarizar (morfológicoGradiente, 0,03);
SE = strel ('quadrado', 2);
máscara = imclose (máscara, SE);
máscara = imfill (máscara, 'buracos');
máscara = bwareafilt (máscara, 2); % de controle do número da área mostrada
notMask = ~ mask;
máscara = máscara | bwpropfilt (notMask, 'Area', [-Inf, 5000 - eps (5000)]);
showMaskAsOverlay (0,5, máscara, 'r'); % você tem que baixar app / function showMaskAsOverlay
BW2 = imfill (binaryImage, 'buracos');
nova_imagem = BW2;
nova_imagem (~ máscara) = 0; % inverter fundo e orifícios
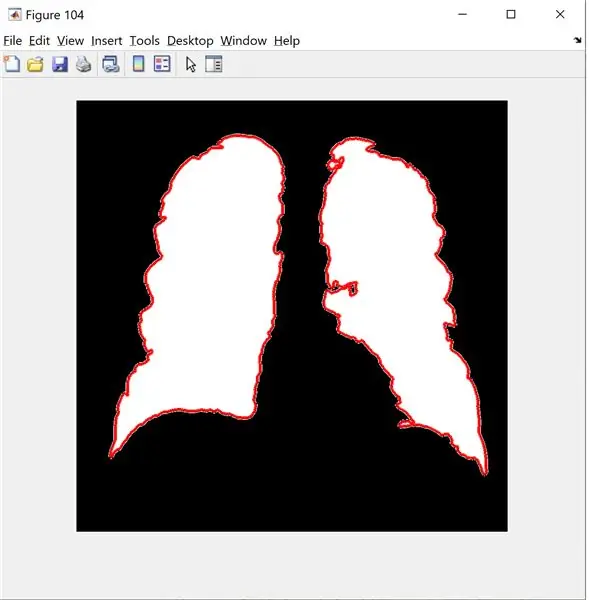
B = limites bw (nova_imagem); % só pode aceitar 2 dimensões
figura (104);
imshow (nova_imagem);
aguentar
fronteiras (B);
fim
Etapa 4: Criando GUI
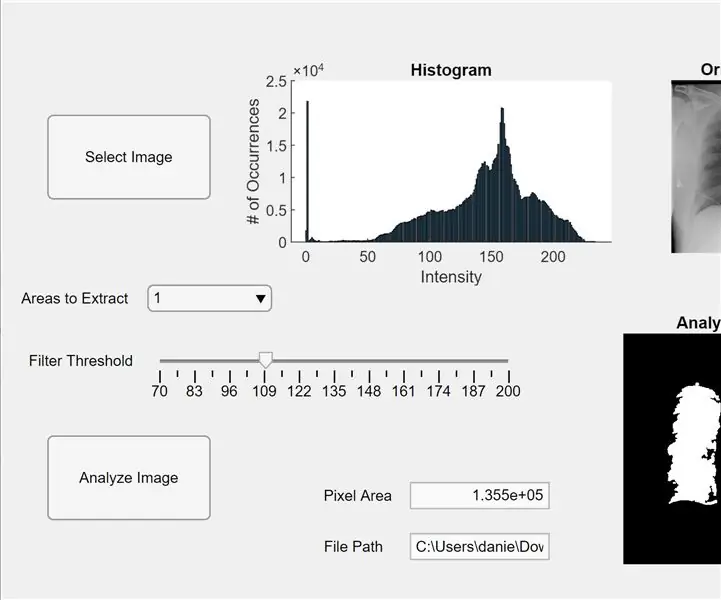
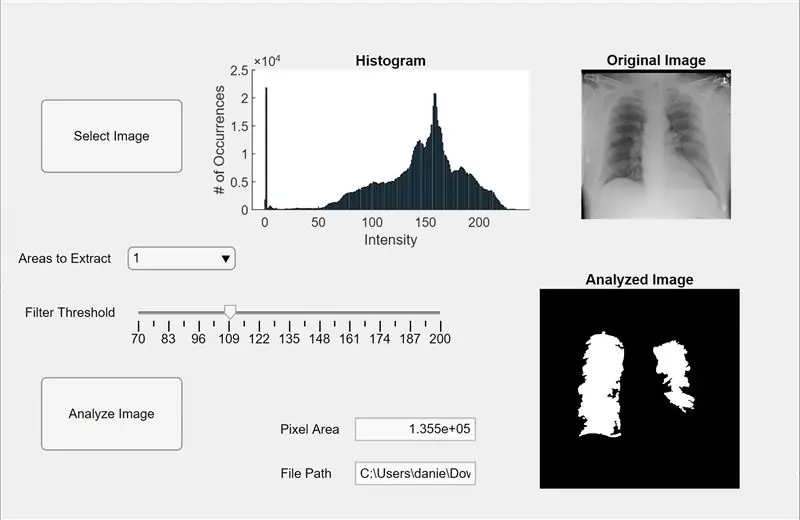
Agora, integramos o código anterior em um aplicativo MATLAB. Abra o App Designer no MATLAB (Novo> Aplicativo). Primeiro, projetamos a interface clicando, segure e arrastando três eixos para a área de trabalho central. Em seguida, clicamos e arrastamos dois botões, um campo de edição (texto), um campo de edição (numérico), um controle deslizante e um menu suspenso. Dois eixos exibirão cada um a pré-visualização e analisarão a imagem, e o terceiro eixo exibirá um histograma de pixels para a pré-visualização da imagem “selecionada”. A caixa do campo de edição (texto) exibirá o caminho do arquivo da imagem selecionada e o campo de edição (numérico) exibirá a área de pixels detectada dos pulmões.
Agora mude da visualização do design para a visualização do código no App Designer. Insira no código o código das propriedades clicando no botão vermelho “Propriedades” com um sinal de adição. Inicialize as propriedades I, threshold e regionToExtract como no código fornecido abaixo. Em seguida, clique com o botão direito em um botão no lado superior direito da área de trabalho (o Component Browser) e vá em Callbacks> Go to … callback. Adicione o código para “function SelectImageButtonPushed (app, event).” Este código permite que você selecione uma imagem para analisar em seu computador usando uigetfile. Depois de selecionar uma imagem, uma imagem de visualização aparecerá sob os eixos acompanhada por um histograma. Em seguida, clique com o botão direito do mouse no outro botão e repita o mesmo procedimento para criar uma função de retorno de chamada.
Adicione o código abaixo de “function AnalyzeImageButtonPushed (app, event)”. Este código irá realizar a contagem de pixels e detecção de blob na imagem de visualização no botão de analisar imagem (aquele que você clicou com o botão direito para este código). Depois de programar os botões, vamos agora programar o controle deslizante e o menu suspenso. Clique com o botão direito no controle deslizante, crie uma função de retorno de chamada e adicione o código abaixo de “function FilterThresholdSliderValueChanged (app, event)” até o final. Isso permite que o controle deslizante ajuste o limite de intensidade de cinza.
Crie uma função de retorno de chamada para o menu suspenso e adicione o código abaixo de “function AreastoExtractDropDownValueChanged (app, evento)” para permitir que o menu suspenso modifique o número de blobs exibidos nos eixos de imagem analisados. Agora, clique em cada entidade no Navegador de componentes e altere suas propriedades de acordo com sua preferência, como alterar os nomes das entidades, remover eixos e alterar a escala. Arraste e solte as entidades do Navegador de componentes na Visualização de design para um layout funcional e fácil de entender. Agora você tem um aplicativo no MATLAB que pode analisar imagens de pulmões por área de pixels!
propriedades (acesso = privado) I = ; % arquivo de imagem
limiar = 257; % limite para binarizar intensidade de cinza
regionToExtract = 2;
fim
função SelectImageButtonPushed (app, evento)
clc; Dir = 'C: / Usuários / danie / Downloads / imagens_004 / imagens'; % define arquivo invariável "prefixo"
[imageExt, path] = uigetfile ('*. png'); % pega a parte variável do nome da imagem
imageName = [Dir filesep imageExt]; % concatenar sequências invariáveis e variáveis
app. I = imread (imageName); % leu a imagem
imshow (app. I, 'pai', app. UIAxes); % mostra a imagem
app. FilePathEditField. Value = path; % mostra o caminho do arquivo de onde a imagem original veio
fim
função AnalyzeImageButtonPushed (app, evento)
originalImage = app. I;
originalImage = wiener2 (app. I, [5 5]); % filtro de remoção de pontos
histograma (app. AxesHistogram, app. I, 256); % exibe o histograma da imagem
a_thresh = originalImage> = app.threshold; % definiu este limite
labelImage = bwlabel (a_thresh);
props = regionprops (a_thresh, 'all');
SortedSolidity = sort ([props. Solidity], 'descer');
SB = solidez classificada (1);
se SB == 1% SB aceita apenas solidez == 1 filtra os ossos
SE = strel ('quadrado', 3);
morphologicalGradient = imsubtract (imdilate (labelImage, SE), imerode (labelImage, SE));
máscara = imbinarizar (morfológicoGradiente, 0,03);
SE = strel ('quadrado', 2);
máscara = imclose (máscara, SE);
máscara = imfill (máscara, 'buracos');
mask = bwareafilt (mask, app.regionsToExtract);
% de controle do número da área mostrada
notMask = ~ mask;
máscara = máscara | bwpropfilt (notMask, 'Area', [-Inf, 5000 - eps (5000)]);
BW2 = imfill (labelImage, 'buracos');
nova_imagem = BW2;
nova_imagem (~ máscara) = 0;
B = limites bw (nova_imagem); % só pode aceitar 2 dimensões imshow (new_image, 'parent', app. UIAxes2);
mantenha (app. UIAxes2, 'ligado');
fronteiras (B);
set (gca, 'YDir', 'reverso');
lungArea = bwarea (nova_imagem);
app. PixelAreaEditField. Value = lungArea;
fim
fim
função FilterThresholdSliderValueChanged (app, evento)
app.threshold = app. FilterThresholdSlider. Value;
fim
função AreastoExtractDropDownValueChanged (app, event) stringNumber = app. AreastoExtractDropDown. Value;
app.regionsToExtract = str2double (stringNumber);
fim
fim
Recomendado:
MATLAB Easy Face Detection: 4 etapas

MATLAB Easy Face Detection: O objetivo principal deste instructables é mostrar o quão fácil será o processamento da imagem. Com a ajuda do MATLABFace, a detecção e rastreamento tem sido um campo de pesquisa importante e ativo, então é por isso que vou explicar como isso pode ser feito com
Como codificar e publicar Matlab 2016b no Word (guia para iniciantes): 4 etapas

Como codificar e publicar Matlab 2016b no Word (guia para iniciantes): Matlab é um programa de linguagem de alto desempenho usado para calcular resultados técnicos. Ele tem a capacidade de integrar recursos visuais, cálculos e programação de uma maneira amigável. Com este programa, o usuário pode publicar problemas e soluções
Controlando Raspberry Pi usando Matlab: 5 etapas

Controlando Raspberry Pi usando Matlab: Ei, este tutorial é sobre como controlar seu framboesa pi sem cabeça usando matlab. Você pode ter que instalar o matlab com a versão mais recente para obter suporte para a placa raspberry pi mais recente
Controlador robótico ROS baseado em Matlab: 9 etapas
ROS Robotic Controller baseado em Matlab: Desde criança, sempre sonhei em ser o Homem de Ferro e ainda o faço. O Homem de Ferro é um daqueles personagens que são realisticamente possíveis e, simplesmente, quero me tornar o Homem de Ferro algum dia, mesmo que as pessoas riam de mim ou digam que é impossível
Aprimoramentos de programação de trem no MATLAB: 6 etapas

Aprimoramentos de programação de trem no MATLAB: Objetivo: O objetivo deste sistema programado é olhar para um Arduino de pequena escala e aplicar a codificação em uma escala maior para aprimorar potencialmente certos recursos de segurança dos sistemas Amtrak Railroad. Para fazer isso, adicionamos um sensor de umidade do solo
